
(Deutsche Fassung, siehe unten)
Dans le cadre de la lutte internationale contre la COVID-19, le département de microbiologie du LNS a apporté son expertise pour l’étude de la « Distribution géographique et temporelle des clades du SARS-CoV-2 dans la région européenne de l’OMS ». L’article correspondant a été publié dans Eurosurveillance, une revue internationale spécialisée dans la surveillance, l’épidémiologie, la prévention et le contrôle des maladies infectieuses.
Cette étude est le résultat d’une vaste collaboration en matière de recherche à l’échelle européenne, et s’appuie sur des données soumises à GISAID (Global Initiative on Sharing Avian Influenza Data – L’initiative mondiale sur le partage des données sur la grippe aviaire). Fondée à l’origine pour l’échange rapide de données sur tous les virus de la grippe, GISAID a également été l’une des premières organisations à recueillir des données sur la séquence du SARS-CoV-2 – le virus qui provoque la maladie COVID-19 – et à les mettre rapidement à la disposition de ses utilisateurs. Du côté du LNS, nos collègues Guillaume Fournier, du service de virologie et de sérologie, et Joël Mossong, Catherine Ragimbeau et Anke Wienecke-Baldacchino, du service d’épidémiologie et de génomique microbienne, ont participé au projet. Sur une période allant de mars à juin 2020, ils ont fourni au GISAID des données de séquençage du génome complet du SARS-CoV-2 pour le Luxembourg.
Séquençage et diffusion des données
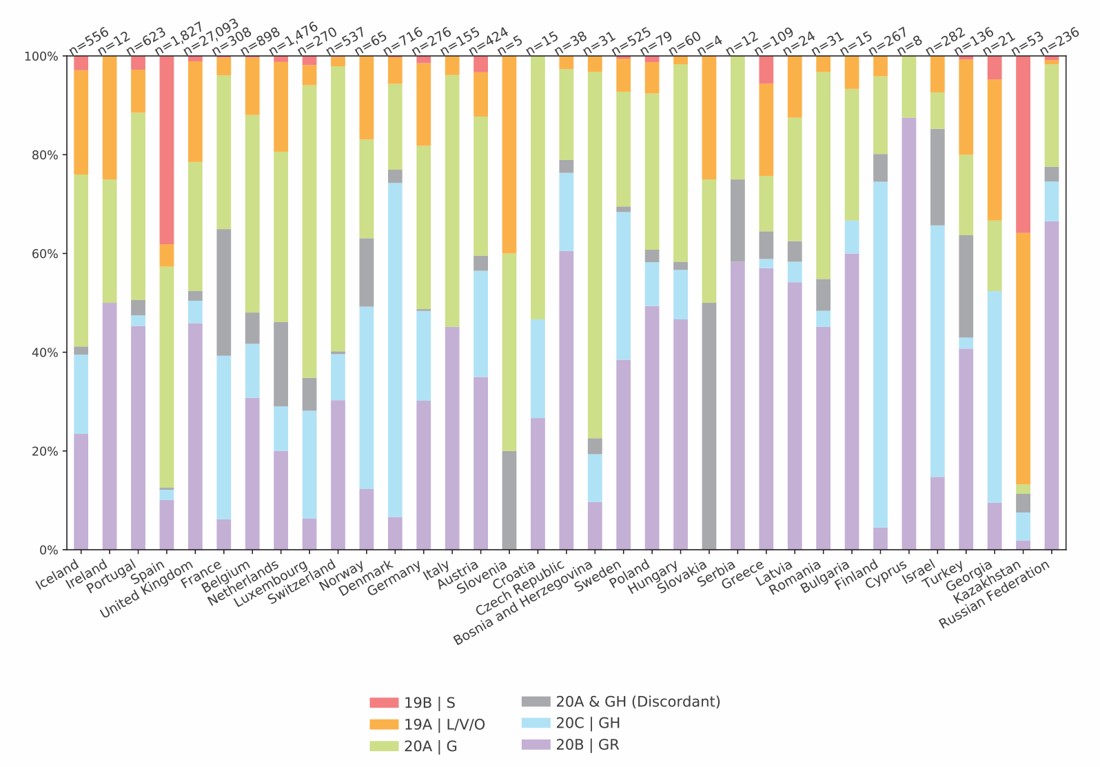
Le but de l’étude était de montrer la répartition des clades génétiques du SARS-CoV-2 au fil du temps et entre les pays, et de définir les objectifs potentiels de la surveillance génomique. Trois systèmes de nomenclature génomique disponibles pour le SARS-CoV-2 ont été appliqués à toutes les données de séquences de la Région européenne de l’OMS disponibles pendant la pandémie de COVID-19 jusqu’au 10 juillet 2020. Nous avons réussi à mettre rapidement en place le séquençage du virus, et avons pu contribuer à hauteur de 271 séquences grâce à un pipeline de séquençage du SARS-CoV-2 systématique et rapide. Le temps de traitement moyen était de seulement 23 jours, contre 32 jours en moyenne dans les autres pays européens. Cela nous a permis de mettre très rapidement les génomes du SARS-CoV-2 présents au Luxembourg à la disposition des archives du GISAID-EpiCoV, ce qui permet une surveillance en temps réel. L’article souligne également l’importance du séquençage en temps réel et de la diffusion des données en situation de pandémie, et fournit une comparaison des nomenclatures, dans le but de jeter les bases de la future surveillance génomique européenne du SARS-CoV-2.
De la recherche polyvalente également au Luxembourg même
Le projet démontre également le rôle de premier plan que joue le LNS dans la lutte contre COVID-19 au Luxembourg et dans les environs. Dans le cas présent, nous avons contribué à un projet de recherche international en apportant les connaissances de notre pays. En outre, nous mettons l’expertise internationale de notre équipe au service du pays et de sa population. En collaboration avec le FNR, nous avons récemment réalisé une étude très intéressante sur le développement des contacts sociaux au Luxembourg pendant et après le confinement. Ce sujet en partie sociologique montre également dans quelle mesure des missions de recherche pertinentes définissent notre rôle en tant que laboratoire.
Lien vers l’article: https://www.eurosurveillance.org/content/10.2807/1560-7917.ES.2020.25.32.2001410
*****
Im Rahmen des weltweiten Kampfes gegen COVID-19 hat das Mikrobiologie Department des LNS seine Expertise in eine Untersuchung zur « geografischen und zeitlichen Verbreitung der SARS-CoV-2-Kladen in der Europäischen Region der WHO » mit eingebracht. Der entsprechende Artikel wurde in Eurosurveillance veröffentlicht, einer internationalen Fachzeitschrift, die auf die Themen Beobachtung, Epidemiologie, Prävention und Kontrolle von Infektionskrankheiten spezialisiert ist.
Die Studie ist das Ergebnis einer umfassenden europaweiten Forschungszusammenarbeit, die auf der Bereitstellung von Daten an GISAID (Global Initiative on Sharing Avian Influenza Data) aufbaut. Ursprünglich für den schnellen Austausch von Daten zu sämtlichen Influenzaviren gegründet, war GISAID auch eine der ersten Organisationen, die Sequenzdaten für SARS-CoV-2 – das Virus welches die Krankheit COVID19 verursacht – erfasst und ihren Nutzern schnell zur Verfügung gestellt hat.
Seitens des LNS waren unsere Kollegen Guillaume Fournier von der Abteilung Virologie und Serologie sowie Joël Mossong, Catherine Ragimbeau und Anke Wienecke-Baldacchino von des Services Epidemiologie und mikrobielle Genomik an dem Projekt beteiligt. Von März bis Juni 2020 stellten sie GISAID die Daten von kompletten SARS-CoV-2-Genomen für Luxemburg zur Verfügung.
Sequenzierung und Datenverbreitung
Ziel der Studie war es, die zeitliche bzw. geografische Verbreitung der genetischen Kladen von SARS-CoV-2 zu untersuchen und somit die Herangehensweise einer künftigen genomischen Überwachung zu definieren. Drei verfügbare genomische Nomenklatursysteme für SARS-CoV-2 wurden auf alle Sequenzdaten aus der Europäischen Region der WHO angewandt, die während der COVID-19-Pandemie und bis zum 10. Juli 2020 verfügbar waren. Wir haben die Sequenzierung des Virus sehr schnell aufgesetzt. Durch den schnellen und effizienten Aufbau einer systematischen und schnellen Sequenzierungspipeline für SARS-CoV-2 konnten wir 271 Sequenzen beisteuern. Die durchschnittliche Bearbeitungszeit betrug dabei 23 Tage, verglichen mit durchschnittlich 32 Tagen in anderen europäischen Ländern. Dies führte wiederum dazu, dass wir die in Luxemburg verbreiteten SARS-CoV-2 Genome sehr schnell dem GISAID-EpiCoV-Archiv zur Verfügung stellen konnte, was einem Echtzeit-Monitoring nahekommt. Der Artikel unterstreicht daher auch die Bedeutung der Echtzeit-Sequenzierung und Datenverbreitung in einer Pandemie-Situation und bietet einen Vergleich der Nomenklaturen mit dem Ziel, eine Grundlage für die künftige genomische Überwachung von SARS-CoV-2 in Europa zu schaffen.
Vielfältige Forschung auch in Luxemburg selbst
Das Projekt zeigt auch die generell führende Rolle, die das LNS im Kampf gegen COVID-19 in und um Luxemburg spielt. Wir haben hier mit der Kenntnis unseres Landes zu einem internationalen Forschungsprojekt beigetragen. Umgekehrt stellen wir das Fachwissen unseres hochinternationalen Teams in den Dienst von Landes und Leuten. So haben wir ja auch gemeinsam mit dem FNR eine hochinteressante Studie über die Entwicklung der sozialen Kontakte in Luxemburg während und nach dem Lockdown durchgeführt. Dieses partiell soziologische Thema zeigt auch, wie breit wir als Laboratorium aufgestellt sind, wenn es um relevante Forschung geht.
Link zum Artikel: https://www.eurosurveillance.org/content/10.2807/1560-7917.ES.2020.25.32.2001410